What is MOCCS?
MOCCS(Motif Centrality Analysis of ChIP-Seq)は,__様々なChIP-Seqのデータから転写因子が認識する配列を正確に抽出できる__新しい解析手法です.
既存のモチーフ探索ツールが位置特異的重み行列(Position weight matrix)など要約された情報を抽出するのに対し,MOCCSは個々の配列がゲノム全体でどの程度認識されているかを評価します.
MOCCSのソフトウェアはGitHubにて公開しています。
https://github.com/yuifu/moccs
私たちはMOCCSを開発し,時計タンパク質CLOCKのChIP-Seqデータに適用し,以下の論文に報告しました.
- Hikari Yoshitane*, Haruka Ozaki*, Hideki Terajima*, Ngoc-Hien Du, Yutaka Suzuki, Taihei Fujimori, Naoki Kosaka, Shigeki Shimba, Sumio Sugano, Toshihisa Takagi, Wataru Iwasaki#, and Yoshitaka Fukada# CLOCK-controlled polyphonic regulations of circadian rhythms through canonical and non-canonical E-boxes. Molecular and Cellular Biology, 34(10):1776 (2014)
- 論文全体の説明
- * Equal contributions, # Corresponding authors
Methods overview
一般に,ChIP-Seqによって決定された転写因子結合サイト周辺での既知の認識配列の出現頻度を調べると,結合サイトを中心としたピーク様の分布を示します(図1).MOCCSはこの性質を転写因子の認識配列の抽出に利用します.すなわち,転写因子結合サイト周辺での長さkの塩基配列(k-mer)の出現頻度がピーク様の分布となる配列を認識配列とし,全てのk-merの中から認識配列を列挙するというのがMOCCSのアイデアです.
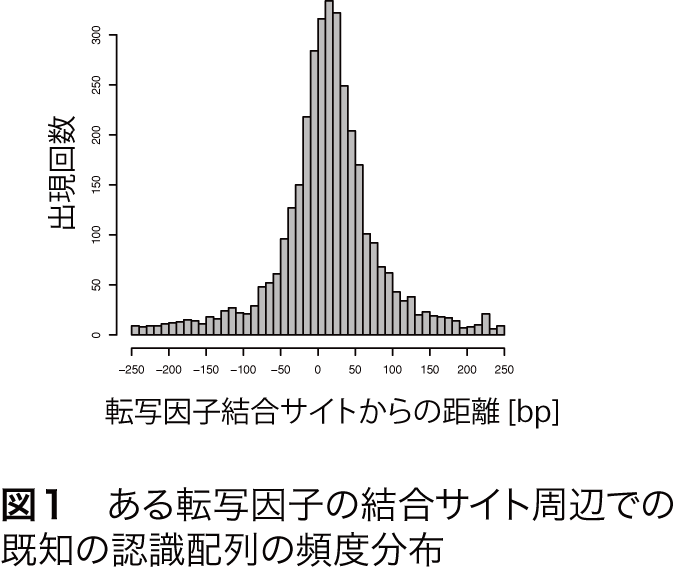
このピーク様の分布の鋭さを定量するために,まず,結合部位の中心からの距離を横軸に取り,累積の出現回数を縦軸に相対値としてプロットします(図2).元の分布がピーク様の分布を示す場合,この曲線は上に凸の形になります(図2,赤線).一方,ある配列が転写因子結合サイトの周辺にランダムに出現する場合は図2の青線のようになります.つまり,頻度分布のピークが鋭いほど,図2の赤線は青線から離れる形になります.この赤線と青線で囲まれた領域の面積をAUC(Area under curve)として定量すると,明確なピーク様分布を示す場合,AUCは大きくなることが分かります(図2).
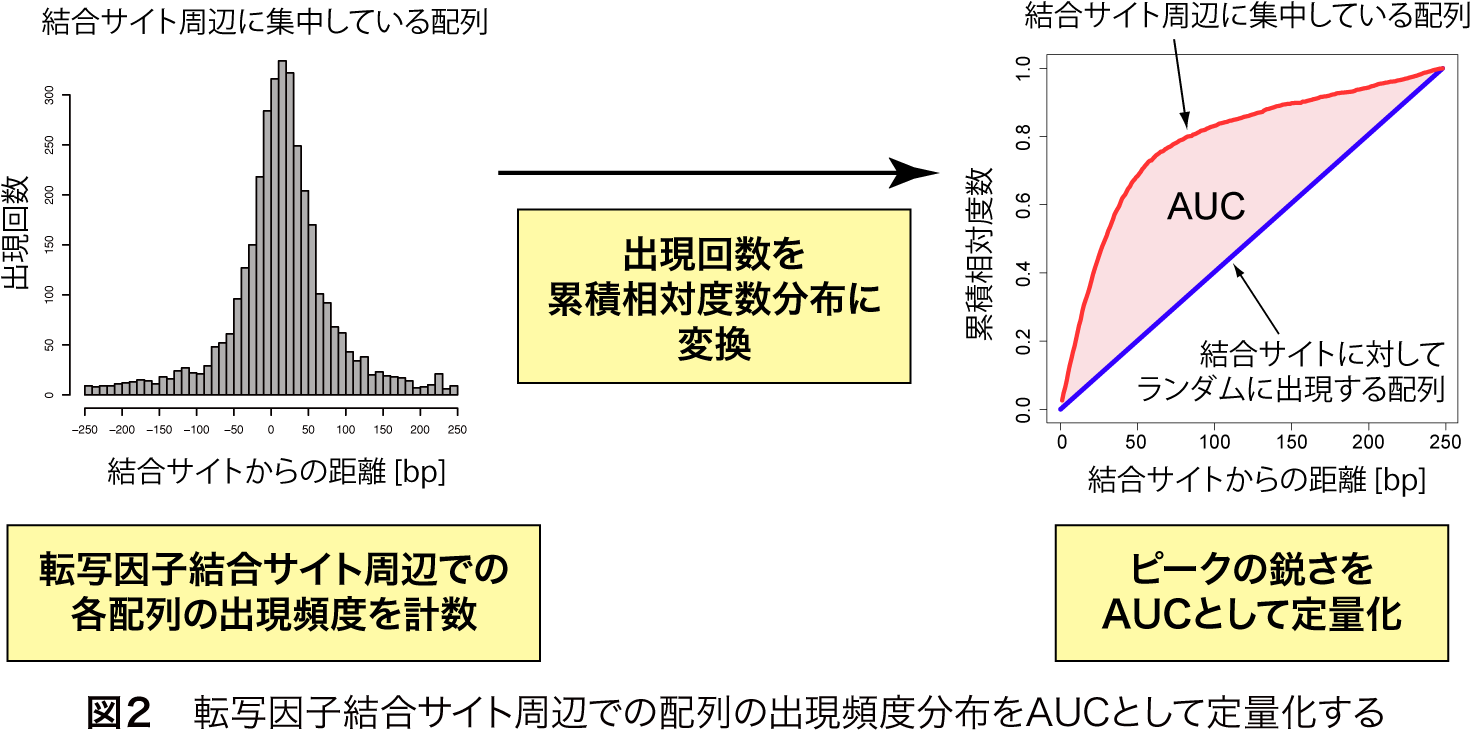
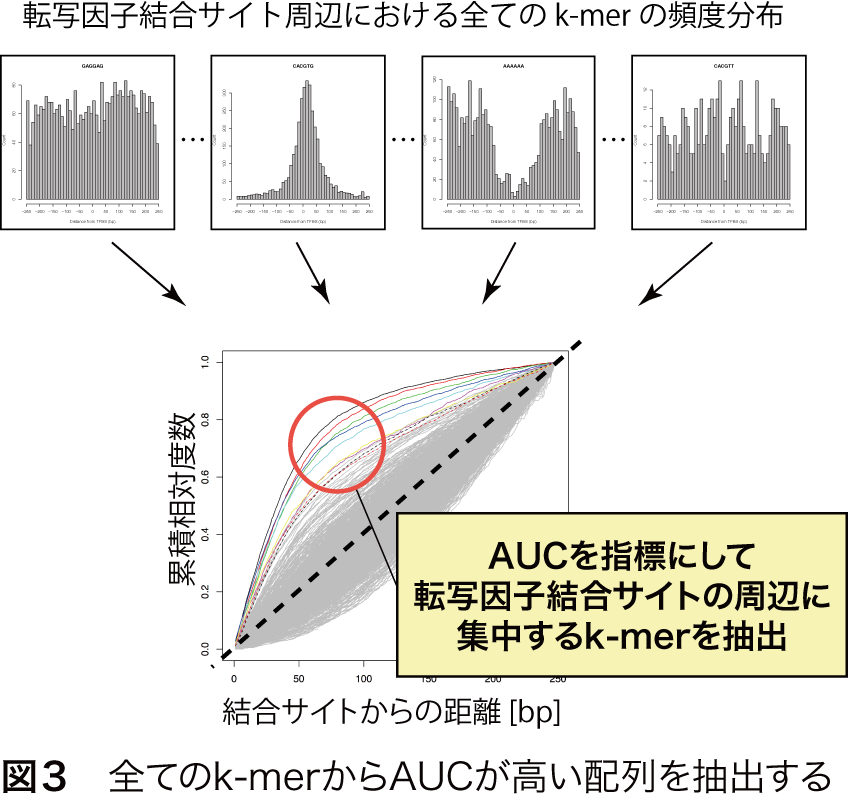
MOCCSは,全てのk-merについて,転写因子結合サイト周辺の頻度分布を調べ,AUCを計算し,AUCが閾値より高いk-merを列挙することで,転写因子の認識配列として抽出することができます(図3).
Use case: CLOCK ChIP-Seq analysis
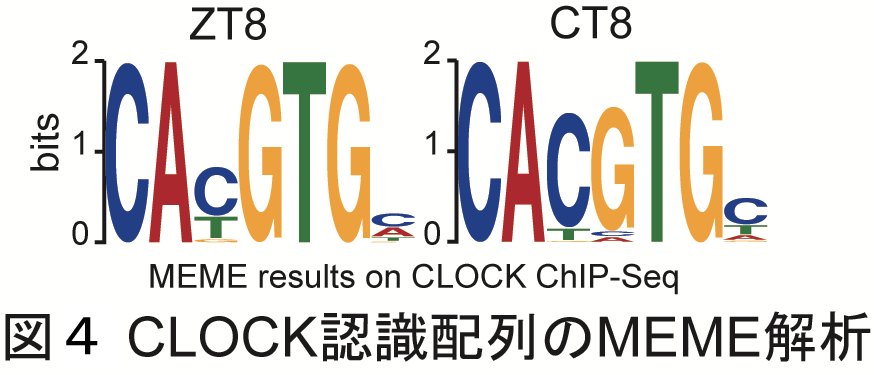
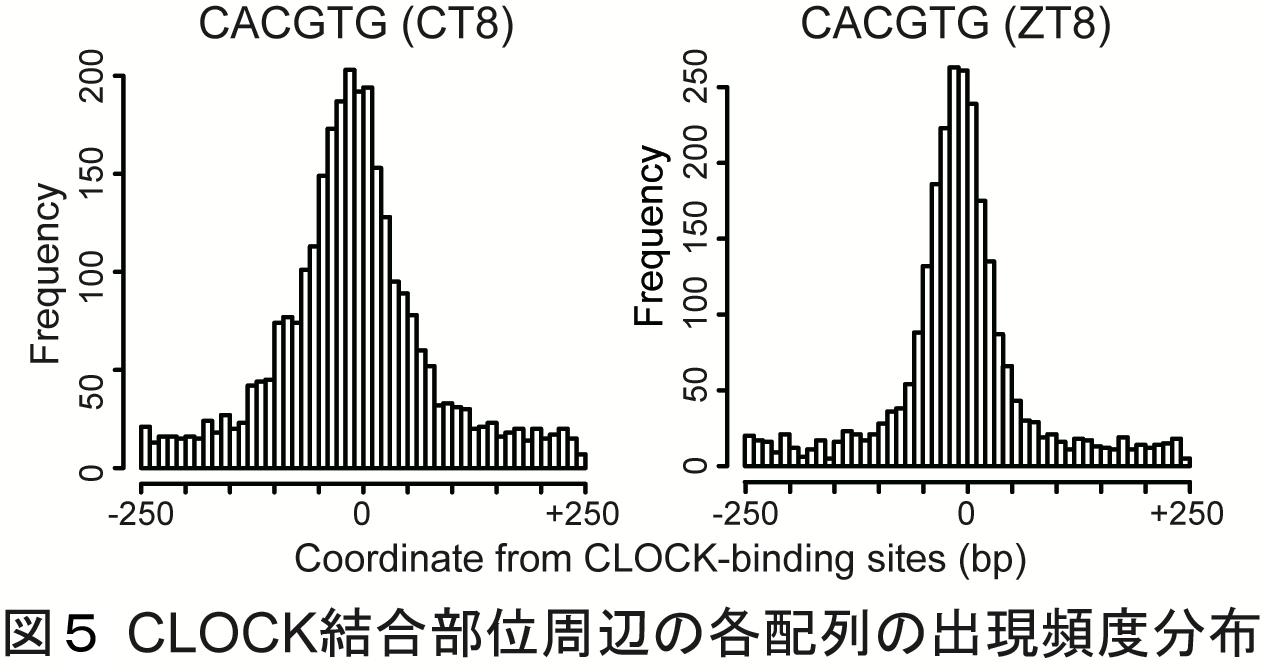
### Distribution of known CLOCK-bidning motifs around CLOCK binding sites まず,CLOCK ChIP-Seqデータからのピーク検出により,約8,000のCLOCK結合サイトを決定しました.この結合サイト周辺±250 bpの配列を解析に使用しました.MEMEによるモチーフ探索をおこなったところ.先行研究の通り,CLOCKの既知の認識配列であるCACGTGが抽出されました(図4).また,CLOCK結合サイト周辺におけるCACGTGの分布をみるとピーク様の分布を示すことが分かります(図5).
MOCCS analysis
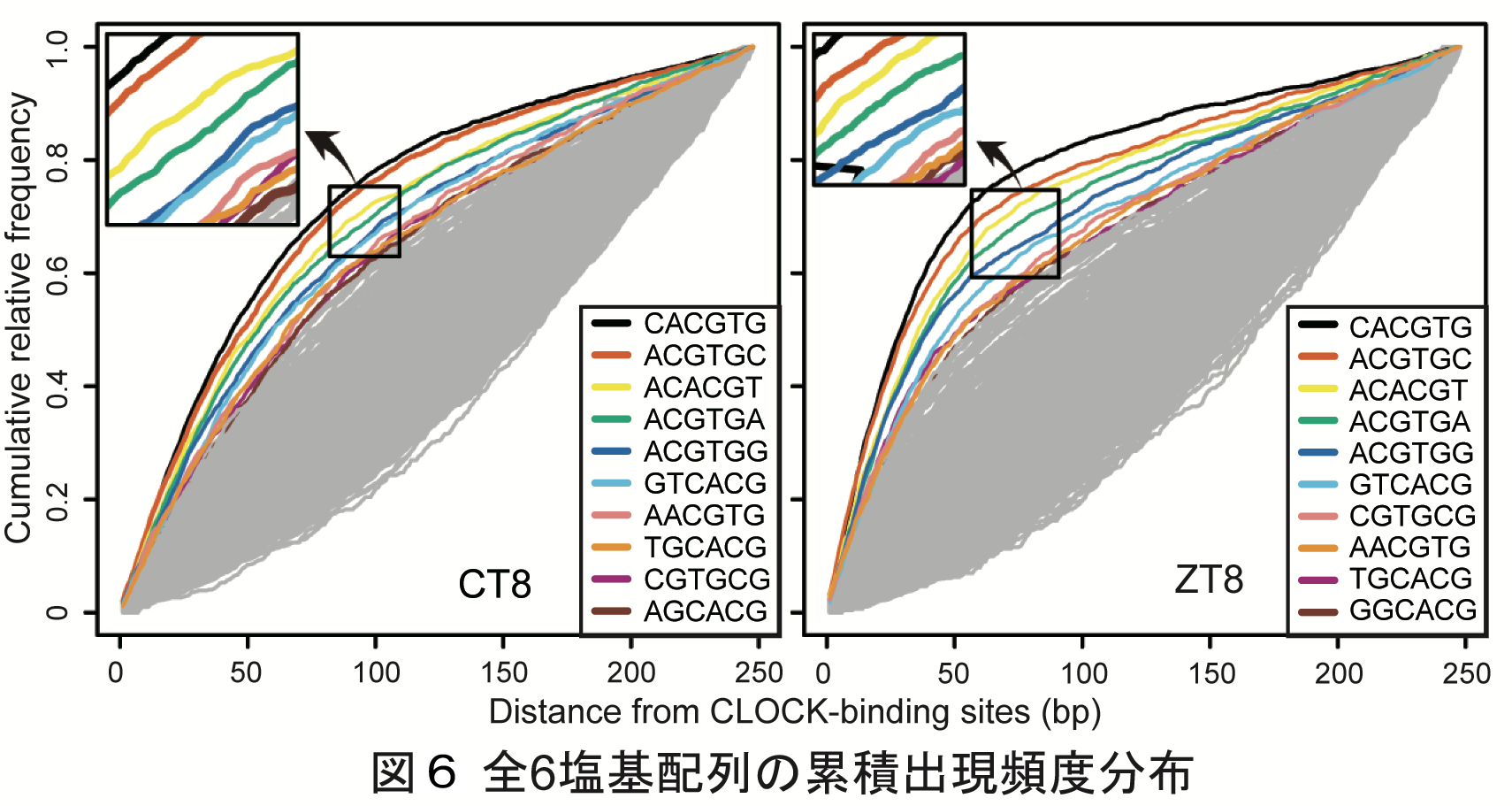
この研究では,CLOCKの典型的な認識モチーフであるCACGTGに類似した配列がどの程度ゲノム全体で使用されているかに興味があったため,探索する塩基配列の長さは6 bpとしました.また,CLOCKは遺伝子の転写開始部位の近傍に結合する傾向を示したため,マウスの全遺伝子のプロモータ配列(TSS上流501 bp)を用いて全ての6-merについてAUCを計算し,その分散の値を用いてAUCを正規化しました.
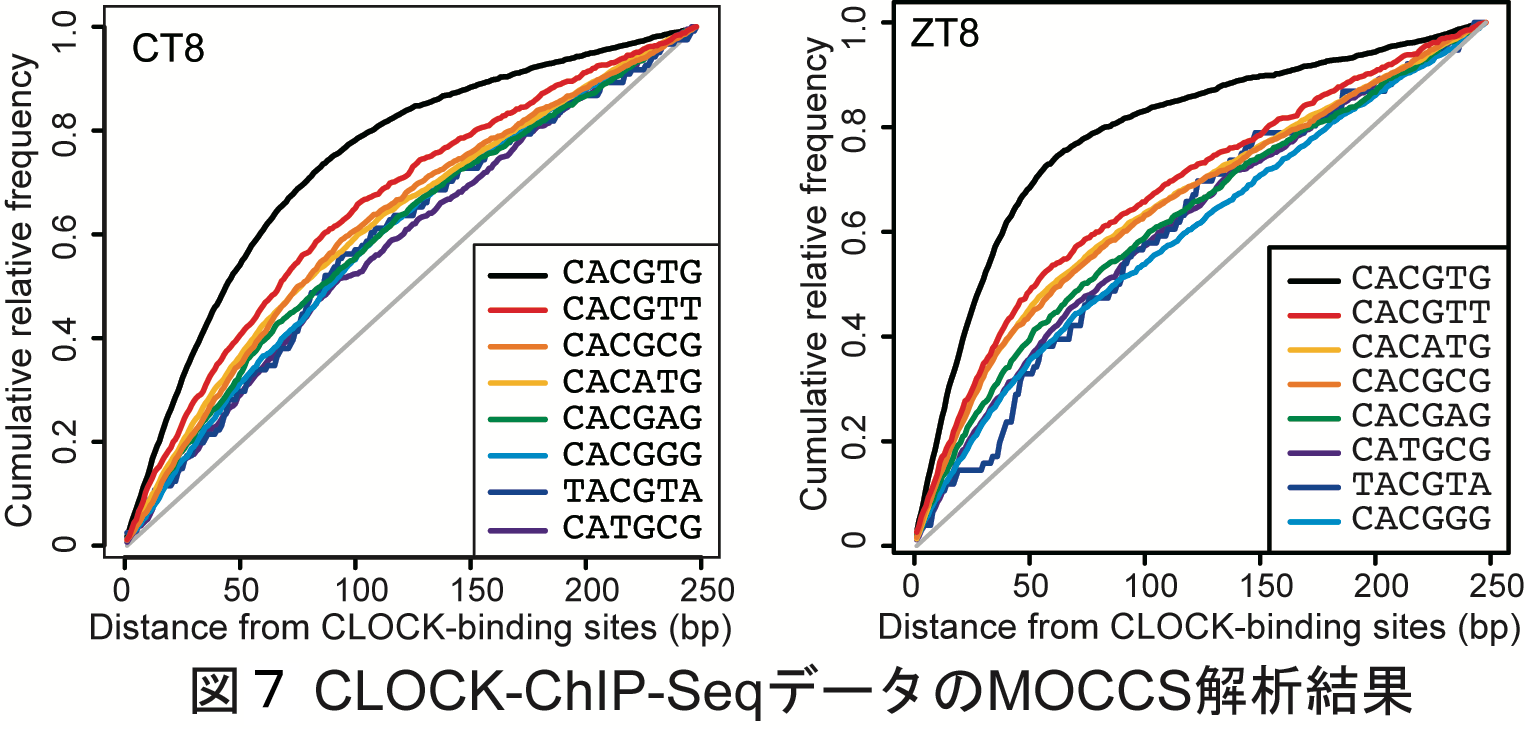
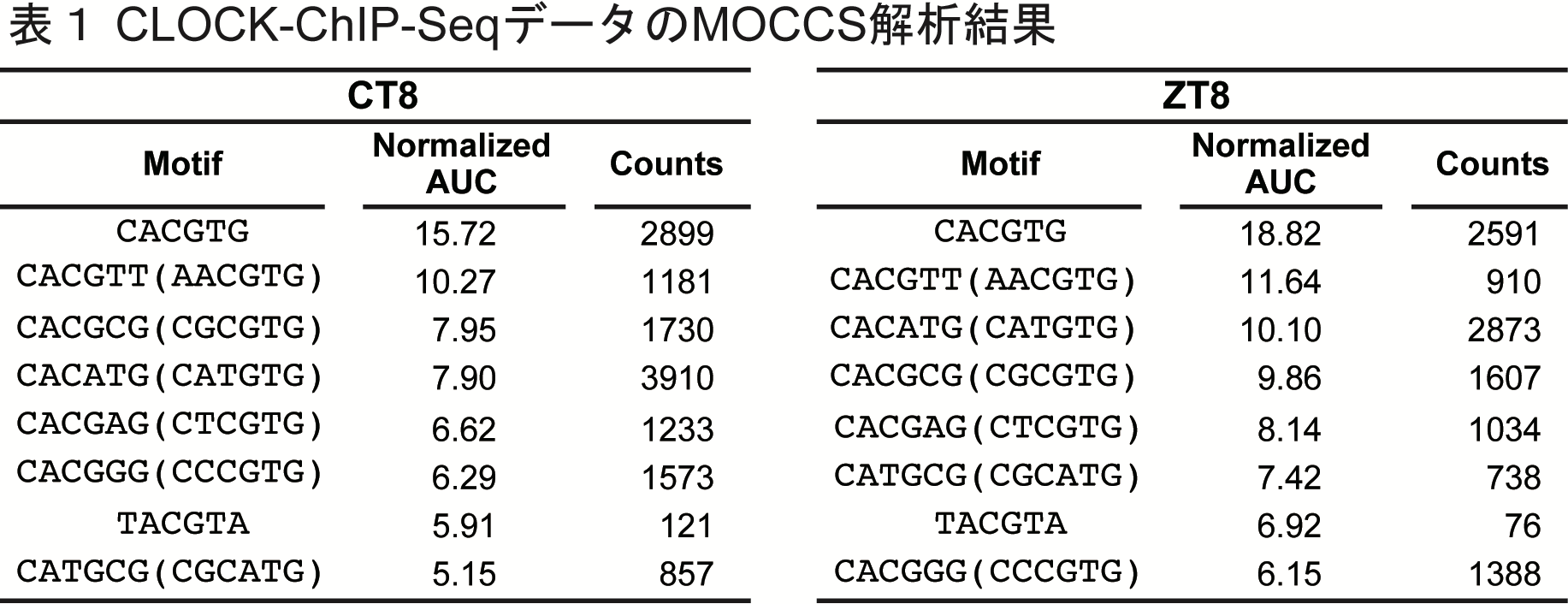
AUCの計算結果をそのまま出力すると,上位3配列はCACGTG,ACGTGC,ACACGTでした(図6).しかし,ACGTGCおよびACACGTはCACGTGから一塩基ずれた配列であるため,このようにすでにAUCで上位にある配列と重なりのある配列は除去しました.最後に,正規化したAUCが5以上という基準を用いて,CLOCK認識配列を抽出しました(図7,表1).
Results and Discussions
MOCCSの結果,AUCが最も高かったのはCACGTGでした(表1).これは.CACGTGが最もよく使用される認識配列であることを示していて,MOCCSが先行研究の知見を再現できたと考えられます.また,CACG[ACGT]Gが全て抽出されたことから,ゲノム全体のレベルでは5番目(2番目)の位置は塩基についてあまり選択的でないことが分かりました.さらに,これまで知られていなかった,CACGTGと2塩基異なる配列が有意な認識配列として見つかりました.
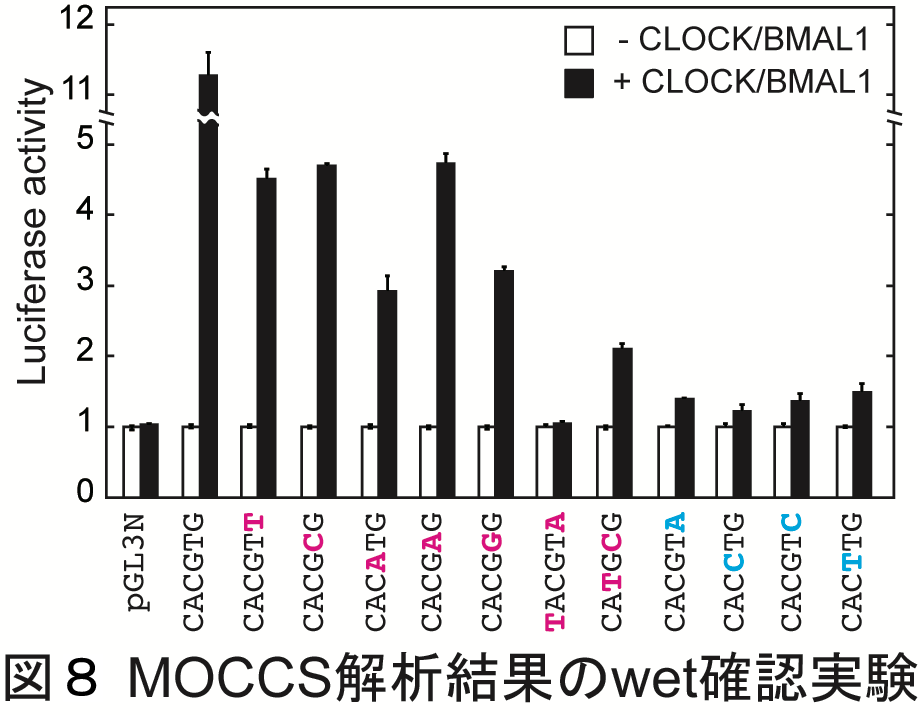
MOCCSで抽出された認識配列が実際に機能を示すかを確かめるため,CLOCK依存的な転写活性化の誘導の有無をルシフェラーゼアッセイにより調べました.その結果,表1のほぼ全ての配列において機能が確認されました(図8).これは,MOCCSが実際に機能する認識配列を正確に予測できたことを表しており,MOCCSの有用性を示すものです.唯一機能を示さなかったTACGTAについては,出現回数が少ないことによってAUCの値がノイズの影響を受けた可能性が考えられます.
Contact
MOCCSは様々なChIP-Seqデータから有意なDNA結合タンパク質の認識配列を列挙することができる手法です.興味のある方は haruka.ozaki AT riken.jp までメールにてご連絡ください.